한국 연안에 서식하는 문절망둑의 지리적 분포와 유전적 거리
ⒸThe Korean Environmental Sciences Society. All rights reserved.
This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
A total of 64 individuals of Acanthogobius flavimanus, which inhabit the coast of Korea, were collected from 8 regions from July to August 2023. A haplotype network and a phylogenetic tree were created. The genomic DNA of the target fish species was compared and analyzed with the genomic DNA of four regions in Japan downloaded from the National Center for Biotechnology Information (NCBI). In the haplotype network of Acanthogoboius flavimanus, Eocheong-do (EC) and Goseong (MAJ) exhibited low genetic similarity with other regions in Korea and Japan. The Phylogenetic tree showed that the population of MAJ exhibited differences in genetic structure compared to populations in other regions of Korea and Japan, indicating a distant relationship. Most marine organisms are known to migrate and spread via ocean currents, which is the most crucial factor promoting gene flow through larvae between populations. The haplotype of Acanthogobius flavimanus in MAJ differs from the haplotypes in Korea and Japan. The population in MAJ is believed to have limited genetic exchange due to the North Korea Cold Currents. We identified haplotype patterns based on the geographical distribution of Acanthogobius flavimanus off the coast of Korea and inferred that ocean currents have some influence on genetic distances.
Keywords:
Acanthogobius flavimanus, Geographic distribution, Genetic distance, Ocean current1. 서 론
망둑어목(Gobiiformes) 어류는 세계적으로 8과 321속 2,167종으로 전 세계 어류의 8 ~ 9%를 차지하고 있다(Moyle and Cech, 2004; Nelson, 2016). 망둑어목 중 망둑어과(Gobiidae)는 약 189속 1,359종이 있고(Nelson, 2016), 국내에는 44속 86종이 연근해와 기수역, 하천에 분포하고 있다(NIBR, 2023).
문절망둑(Acanthogobius flavimanus)은 한반도를 비롯하여 일본열도, 연해주, 발해해, 황해 등 북동아시아가 원 서식지로(Akihito et al., 2002) 선박 평형수(Ballast water)에 의해 캘리포니아와 오스트레일리아 해역까지 확산되었다(Hirase et al., 2017). 최근에 유생 및 미성어가 북베트남의 Ka Long 기수역에서 관찰되었다(Ta et al., 2022). 문절망둑의 확산은 자어기가 길어질수록 더 광범위하고, 유전적 분화가 낮아질 것으로 예상된다(Burton, 1983; Bohonak, 1999).
망둑어과 어류는 연안 생태계의 먹이사슬에서 매우 중요한 위치를 차지하고 있어 국내 및 국외에서 다양한 연구가 진행되고 있다. 또한, 문절망둑은 낚시 및 식용에 이용되는 주요 종이며, 레저용 또는 상업용으로 가치가 높은 종이다(Choi, 2011; Hirase et al., 2017).
문절망둑과 관련한 연구는 국외에서 망둑어류에 대한 유전학 및 형태적 분석(Song et al., 2010a; Song et al., 2010b; Zhicheng S. et al., 2020), 외래종 기원 및 생태(Neilson and Wilson, 2005; Moore et al., 2008; Hirase et al., 2017), 성장과 성숙에 대한 연구(Suzuki et al., 1989; Kuno and Takita, 1997) 등이 다양하게 진행되었다.
국내에서는 분류, 생태 및 골학적 특성 연구(Lee, 1992; Choi et al., 1996; Lee, 2001)와 망둑어류의 분류학적 재검토 및 종 목록 변화 연구가 있다(Kim et al., 1987; Lee, 2013). 그러나, 망둑어과 어류의 유전적 특성과 지리적 분포에 따른 분자계통학적 연구는 미흡하다. 따라서, 본 연구에서는 우리나라 각 연안에 서식하는 문절망둑 개체군의 유전적 거리와 일본 연안에 분포하는 개체군과의 분자계통학적 관계를 파악하고자 한다.
2. 재료 및 방법
2.1. 대상 종 채집
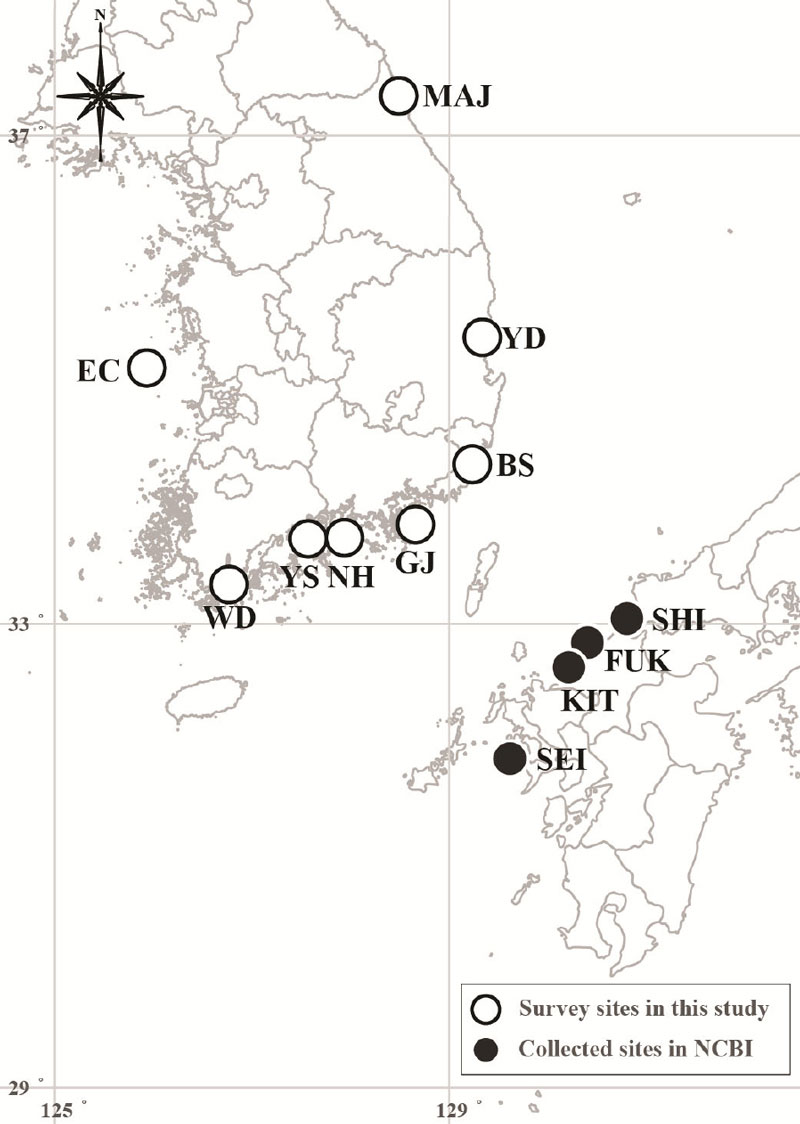
2023년 7월부터 8월까지 우리나라 전 연안에서 투망(15 × 15 ㎜)과 족대(5 × 5 ㎜)를 이용하여 대상 어종을 채집하였다. 또한, 일본의 DNA 염기서열은 미국 국립생물정보센터(National Center for Biotechnology Information, NCBI)에서 제공하는 자료를 이용하였다(Fig. 1, Table 1).
유전자 분석용 시료는 대상어의 근육 조직 또는 어체 전체를 95% 에탄올에 고정하여 급속 냉동고(-70℃)에 보관하였다. 보관된 시료는 DNA 분해를 최소화하기 위해 2주 이내에 게놈 DNA 추출물을 분석하였다.
2.2. 미토콘드리아 DNA 분석
유전자 분석용 채취 시료에서 미토콘드리아 DNA를 Silica Magnatic Bead 방법으로 추출하였다. 본 연구에서는 미토콘드리아 DNA 영역 중 Cytochrome c oxydase subunit Ⅰ (COⅠ)와 D-loop (Control region) 영역을 이용하였다. 추출한 DNA는 Hirase et al. (2017)에서 개발한 유전자 primer COⅠ (Forward: 5ʹ-ATGGGGCTACAATCCACCAC-3ʹ, Reverse: 5ʹ-AGGTGAAGTATGCYCGTGTG-3ʹ)과 D-loop (Forward: 5ʹ-TCCCATCTCTAGCTTCCCAAA-3ʹ, Reverse: 5ʹ-AGACTTTCCTGTTTCCGGGG-3ʹ)를 사용하여 증폭하였다.
대상 어류의 게놈 DNA 2 ㎕, dH2O 15.1 ㎕, 10×Reaction Buffer 2 ㎕, dNTP 0.5 ㎕(각 150 nM), Primer(F/R) 각 0.1 ㎕, Hot-start Taq polymerase 0.2 ㎕ (1 unit)를 순차적으로 첨가하여 총 20 ㎕로 혼합하였다. 혼합된 PCR 반응 용액은 ProFlex™ PCR system (Thermo Fisher Scientific Corporation, USA)을 이용하여 초기 열변성 95℃ 5분 이후, 95℃ 20초, 58℃ 20초, 72℃ 35초를 32회 반복하였다. 최종신장 반응은 75℃ 5분간 진행한 다음 4℃로 유지하였고, 2% 아가로스 젤에서 전기영동하여 PCR products를 확인하였다.
최종 PCR products는 Silica magnetic bead로 dye terminator kit (BigDye™)로 mtDNA를 정제하였다. 정제된 mtDNA 염기서열은 본 연구에 이용한 프라이머 쌍을 Macrogen Inc. (Seoul, Korea)에 제공하여 양방향으로 염기서열을 결정하였다. Geneious Prime ver. 2023.2.1. (Raco et al., 2023)을 이용하여 Alignment 및 Assemble하였다.
2.3. 데이터 분석
문절망둑의 mtDNA 분석 및 수동 수정을 위해 Geneious Prime을 사용하였다. 정렬된 염기서열은 PopART ver. 1.7 (Alvi et al., 2023)을 이용하여 haplotype을 확인하였고, 각 지점의 단상형 분포를 도식화하기 위해 TCS Network로 분석하였다.
본 연구 대상 어류 64개체를 대상으로 분자계통 분석을 수행하였다. Outgroup은 근연종인 A. stimothomus로 설정하였으며, 분자계통도 작성을 위해 IQ-TREE ver. 2.2.2.6 (Zhang et al., 2020)를 이용하여 Maximum Likelihood(ML)를 분석하였고, bootstrap은 1,000번으로 설정하였다. Bayesian Information Criterion (BIC)는 종 및 사용한 유전자에 따라 다르게 적용하였으며, K2P (COⅠ), HKY + F + G4(D-loop) 모델을 적용하였다. FigTree ver. 1.4.4 (Zhang et al., 2018)로 분자계통도를 도식화하였다.
국내 연안에서 분석한 문절망둑의 DNA 염기서열은 일본(Shimonoseki (SHI), Fukuoka (FUK), Kitakyushu (KIT), Seikai (SEI))의 DNA 염기서열과 비교하였다(Table 2). 일본의 문절망둑 데이터는 D-loop 영역을 각 지점당 2개체씩 총 8개체를 사용하였다.
3. 결 과
3.1. 단상형 네트워크(Haplotype network) 분석 및 지리적 분포
문절망둑의 채집은 우리나라 동·서·남해안의 연안에서 진행되었다. 서해안에서는 어청도(EC)에서 8개체가 채집되었으며, 남해안에서는 완도(WD), 여수(YS), 남해(NH)와 거제(GJ)에서 32개체가 출현하였고, 동해안에서는 부산(BS), 영덕(YD), 고성(MAJ)에서 24개체가 확인되었다. 총 8개 지점에서 64개체(지점당 각 8개체)의 문절망둑을 채집하였으며, 채집한 대상 어류에서 추출한 DNA의 서열 길이는 COⅠ가 921 bp이었고, D-loop는 796 bp이었다.
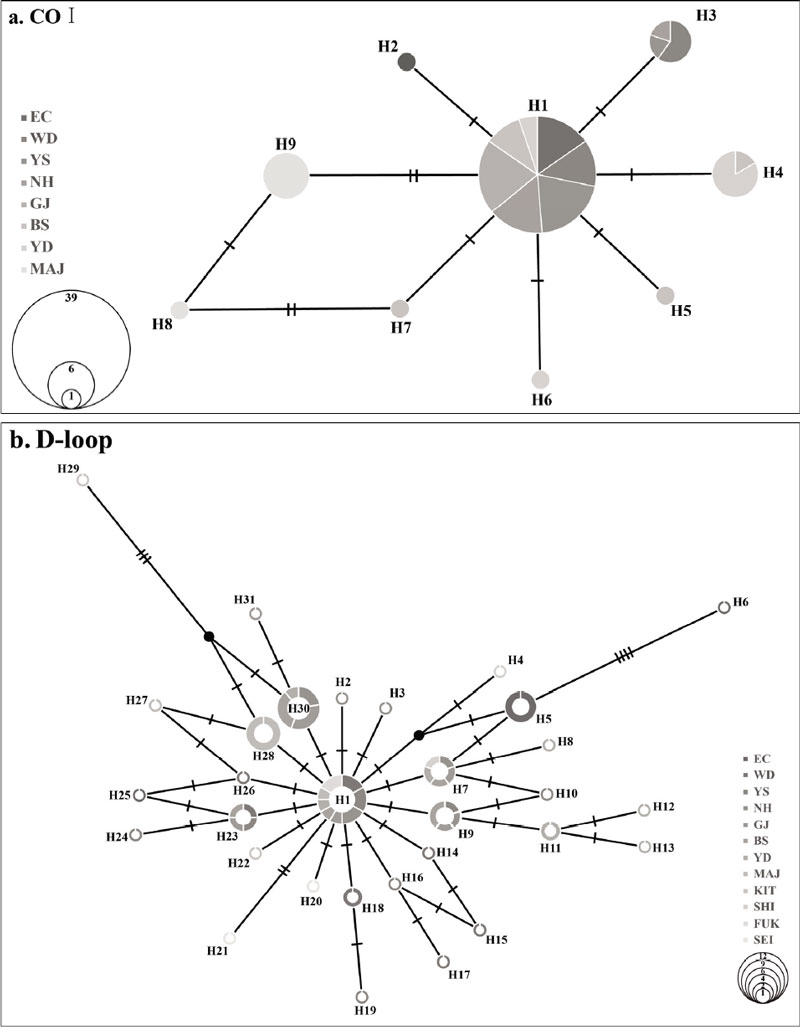
64개체의 국내 표본을 COⅠ로 분석한 결과, 9개의 Haplotype (H1~H9)이었다. 또한, 64개체의 국내 표본과 8개체의 일본 DNA 염기서열을 D-loop로 비교·분석한 결과, 32개의 Haplotype (H1~H32)로 확인되었다. 또한, 분석된 Haplotype network는 모두 별 모양을 형성하였다(Fig. 2).
지리적으로 Haplotype을 분석한 결과, COⅠ 영역에서는 Haplotype 수가 4개 (H1, H4, H5, H7)로 가장 다양하게 나타난 BS에서는 H1이 4개체, H4가 2개체, H5와 H7이 각각 1개체 순으로 분석되었다. YS와 GJ에서는 모두 H1으로 단순한 Haplotype을 나타냈다 (Fig. 4).
D-loop 영역에서 Haplotype 수가 가장 다양하게 나타난 지점은 YS와 YD로 각각 7개로 나타났다. YS에서는 H1이 2개체로 확인되었고, H9와 H10, H16, H19, H23, H24가 각각 1개체로 관찰되었다. YD에서는 H11이 2개체로 가장 높았고, H1과 H7, H8, H12, H13, H30이 각각 1개체로 분석되었다(Fig. 4).
3.2. 계통수(Phylogenetic tree) 및 유전적 거리
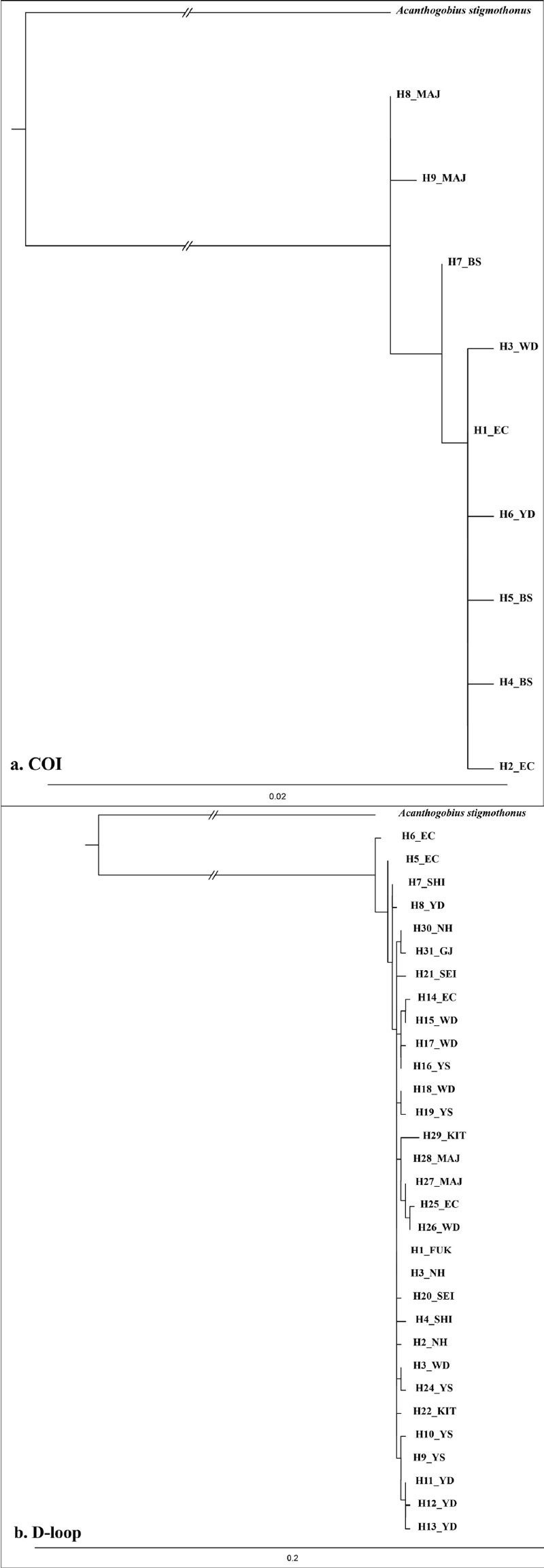
COⅠ 영역의 계통수는 우리나라에서 채집된 개체군을 대상으로 분석하였다(Fig. 3a). MAJ (H8_MAJ, H9_MAJ) 개체군에 비해 BS (H4_BS, H5_BS), EC (H1_EC, H2_EC), YD (H6_YD)과 WD (H3_WD) 개체군 간의 유전적 거리가 유사하거나 짧은 것으로 나타났다.
D-loop 영역의 계통수는 우리나라와 일본의 큐슈 북부 4개 지점의 개체군을 대상으로 분석하였다(Fig. 3b). NH (H18_NH, H19_NH), FUK (H1_FUK), SEI (H16_SEI, H17_SEI, H15_SEI), KIT (H4_KIT)와 SHI (H14_SHI) 지역에 분포하는 개체군 간의 유전적 거리가 가까운 것으로 확인되었다.
유전적 거리는 유전적 동질성(Genetic identity)의 결과값을 이용하였다. COⅠ 영역은 강원도 고성 개체군(0.9955 ~ 0.9989)이 다른 나머지 개체군(0.9978 ~ 0.9989)에 비해 근소한 차이를 보였다. 반면, H8_MAJ Haplotype이 H2_EC와 H3_WD, H4_BS, H5_BS, H6_YD Hplotype과 0.9955로 유전적 거리가 가장 먼 것으로 확인되었다(Table 3).
D-loop 영역에서는 H6_EC Haplotype이 H31_ KIT Hplotype과 0.9818로 유전적 거리가 먼 것으로 확인되었고, H3_NH Haplotype이 H1_FUK와 1.0000으로 유전적 거리가 차이가 거의 없는 것으로 나타났다(Table 4).
4. 고 찰
우리나라와 중국, 일본의 문절망둑에서 추출한 DNA로 Haplotype network와 Phylogenetic tree를 도식화하여 지역적으로 분석하였다.
우성 Haplotype (H1)은 국내 지역 중 MAJ (COⅠ)와 EC (D-loop)를 제외한 모든 지역에서 관찰되었으나, 2개 마커(COⅠ, D-loop) 유형이 모두 별 모양의 Haplotype network로 확인되었다(Fig. 2). 이는 지역적 분리로 인해 해당 개체군이 유전적 분화가 발생한 것으로 Heo et al.(2022)의 결과와 일치하였다.
EC 및 MAJ 개체군은 국내 다른 지역(WD, YS, NH, GJ, BS, YD)과 일본 지역(SHI, FUK, KIT, SEI)에 비해 유연관계가 낮은 것으로 나타났다. 이는 COⅠ 영역에서 H8과 H9의 유형이 MAJ에서만 관찰되었고, D-loop 영역에서는 H5와 H6, H14, H25의 유형이 EC에서만 확인되었다. 또한, 계통수는 MAJ의 개체군이 국내 다른 지역 및 일본 개체군의 유전자 구조가 먼 것으로 확인되었다.
COⅠ 영역의 EC 개체군의 H1은 MAJ 지역을 제외한 모든 지역에서 확인되었다. MAJ 개체군은 H8와 H9의 Haplotype이 확인되었고, 유전적 동일성에서 MAJ 개체군이 다른 지역 개체군보다 먼 거리의 결과값이 나타났다. 이러한 결과로 MAJ 개체군은 국내 개체군에서 파생되거나 유전적 교류로 인해 분화된 개체군이 아닌 다른 지역에서 유입된 개체군으로 판단된다(Table 3, Fig. 2a, 4).
D-loop 영역은 FUK 지역의 H1이 7개 지역(WD, YS, NH, GJ, BS, YD, MAJ)에서 관찰되었으며, EC에 서식하는 개체군과는 다른 Haplotype (H5, H6, H14, H25)을 가지고 있는 것으로 관찰되었다. 유전적 동일성에서는 EC 개체군이 다른 지역의 개체군과 유전적 거리가 먼 것으로 확인되었다. 따라서, EC 개체군은 동·남해안의 개체군이나 일본 개체군이 아닌 지역의 개체군이 유입되거나 문절망둑의 연안성 생태와 도서 지역의 특성으로 개체 간의 유전적 교류가 빈약하여 분화된 개체군으로 판단된다(Fig. 2b, 4).
해류는 해양 생물의 서식 및 이동에 영향을 주는 중요한 메커니즘이고, 개체군 간 유생 교류를 통해 유전자 흐름을 촉진하는 가장 중요한 요인이다(Song et al., 2010 b). 해양 생물은 해류를 통해 최소한의 에너지로 먼 거리를 이동한다. 특히, 유영 능력이 약하거나 없는 부유 생물의 이동에 영향을 주는 것으로 알려져 있다.
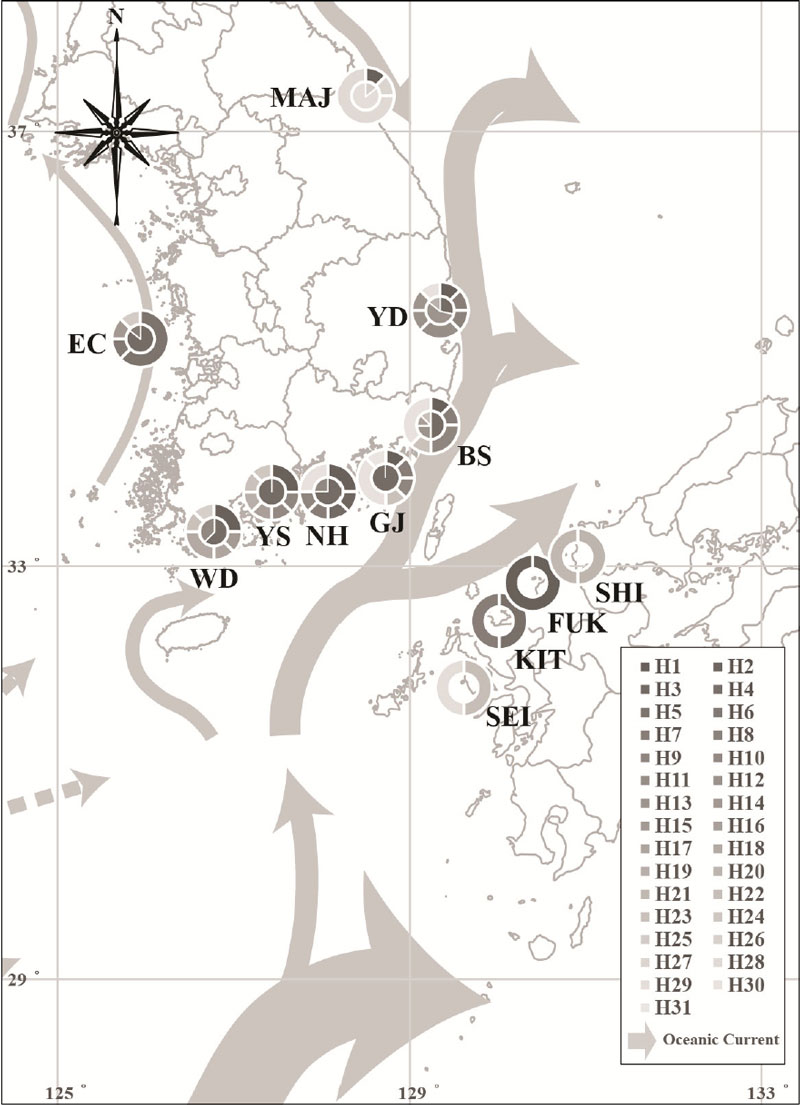
우리나라 주변 해역의 해류는 고온‧고염의 쿠로시오 해류를 통해 제주도, 남해안 및 대한해협을 통하여 서해와 동해로 흐른다. 대마난류의 일부는 서해안으로 북상하고, 나머지는 대한해협을 거쳐 동해로 북상하는 동한난류와 일본 열도를 따라 북상하는 일본연안지류와 외해지류로 구분된다. 동해안의 북쪽 해역은 리만한류가 북한 해역으로 남하하면서 북한한류로 명명되어 남하하고 있다. 또한, 서해에는 서쪽 연안을 따라 흐르는 서한연안류와 산둥 반도를 지나 중국 동쪽 연안을 따라 흐르는 중국연안류가 흐르고 있다(Park et al., 2016)(Fig. 4). 북한한류와 서한연안류는 MAJ 지역의 개체군과 EC 지역의 개체군이 동·남해안 및 일본의 다른 지역(YD, BS, GJ, NH, YS, WD, SHI, FUK, KIT, SEI)의 개체군과의 지리적 분포에 영향을 주어 결과적으로 문절망둑의 유전자 흐름(Gene flow)에 영향이 발생하였을 것으로 사료된다.
MAJ 개체군은 YD부터 WD까지의 개체군과 다른 Haplotype이 나타났으며, 이는 MAJ 지역은 대마난류보다는 북한한류의 영향권에 속하고, YD부터 WD까지는 대마난류의 영향을 받는 지역으로 해류의 영향권이 서로 다름으로 인하여 MAJ 개체군과 이 이외의 다른 지역 개체군은 서로 유전적 교류가 원활하지 않은 것으로 판단된다.
본 연구는 최근 기후 변화에 따른 수온 상승으로 생태계의 위협과 종조성의 변화에 따른 해양 생물의 복원 및 보전에 대한 기초자료로 활용될 수 있을 것이며, 이를 위해서 연안 저서성 어류의 이동과 확산, 종 기원(Origin) 및 유전적 구조, 계통지리학적(Phylogeography) 분석 등의 연구가 추가적으로 필요하다.
5. 결 론
본 연구는 우리나라 연안에 분포하고 있는 문절망둑 개체군의 유전적 거리와 일본 연안에 분포하는 개체군과의 분자계통학적 관계를 파악하였다. 분자계통 분석은 서해안 1지점 8개체, 남해안 4지점 32개체, 동해안 3지점 24개체 총 8지점 64개체를 COⅠ과 D-loop 영역에서 수행하였다. COⅠ 영역은 BS에서 4개의 Haplotype(H1, H4, H5, H7)이 확인되었고, D-loop 영역의 Haplotype은 YS (H1, H9, H10, H16, H19, H23, H24)와 YD (H1, H7, H8, H11, H12, H13, H30)에서 각각 7개로 분석되었다. COⅠ 영역에서 MAJ Haplotype은 다른 지점에 없는 H8과 H9를 가지고 있으며, D-loop 영역에서는 EC Haplotype에서 다른 지점에 속하지 않는 H5와 H6, H14, H25를 가지고 있다.
유전적 동질성 결과에서 COⅠ 영역은 MAJ 지점의 H8_MAJ Haplotype이 다른 지점의 Haplotype (H2_EC, H3_WD, H4_BS, H5_BS, H6_YD)과 0.9955로 유전적 거리가 가장 먼 것으로 확인되었다. D-loop 영역에서는 EC 지점의 H6_EC Haplotype이 H31_KIT Haplotype과 0.9818로 가장 먼 유전적 거리를 나타내었다.
문절망둑은 부유자어기를 가지고 있으며, 유영 능력이 약하거나 없어 이동의 제약이 있을 것으로 판단되고, 이를 극복하기 위해 해류를 통해 최소한의 에너지로 먼 거리를 이동한다. 우리나라 연안의 해류는 서해안에 서한연안류와 중국연안류가 흐르고 있다. 남해안에는 대마난류의 일부가 서해안으로 북상하고, 나머지는 대한해협을 통하여 동해로 흐른다. 동해안에는 북상하는 동한난류와 남하하는 북한한류가 만나고 있다. 이러한 해역별 해류의 특성이 유전자 흐름을 촉진하는 중요한 요인으로 작용하고 있다.
본 연구 조사 지점인 EC 및 MAJ 개체군은 다른 지역의 개체군과 다른 유전자 구조를 가지고 있으며, 이는 서한연안류와 북한해류가 EC 및 MAJ 지역의 개체군에 영향을 주어 문절망둑의 유전자 흐름에 영향이 발생하였을 것으로 사료된다.
References
-
Alvi, M. A., Khalid, A., Ali, R. M. A., Saqib, M., Qamar, W., Li, L., Ahmad, B., Fu, B., Yan, H., Jia, W., 2023, Genetic variation and population structure of fasciola hepatica: An in silico analysis, Parasitol.Res., 122, 2155-2173.
[https://doi.org/10.1007/s00436-023-07917-0]

-
Bohonak, A. J., 1999, Dispersal, gene flow, and population structure, Q. Rev. Biol., 74, 21-45.
[https://doi.org/10.1086/392950]

- Burton, R. S., 1983, Protein polymorphisms and genetic differentiation of marine invertebrate populations, 4, 193-206.
- Choi, Y., 2011, The goby, Jisungsa, Seoul, Korea, 69-101.
- Choi, Y., Kim, I., Ryu, B., Park, J., 1996, Ecology of Synechogobius hasta (pisces: Gobiidae) in the kum river estuary, Korea, 29, 115-123.
- Choi, Y., Kim, J., Park, J., 2021, Marine fishes of Korea, 5th, Kyohak-sa, Seoul, Korea, 460-485.
- Fan, H. Y., Ji, Y. P., Zhang, S. H., Yuan, C. T., Gao, T. X., 2005, Research of fishery biology of the neritic fish Synechogobius ommaturus in the area of the huanghe delta, 35, 733-736.
-
Heo, N., Lomolino, M. V., Watkins Jr, J. E., Yun, S., Weber-Townsend, J., Fernando, D. D., 2022, Evolutionary history of the asplenium scolopendrium complex (aspleniaceae), a relictual fern with a northern pan-temperate disjunct distribution, Biol. J. Linn. Soc., 137, 183-199.
[https://doi.org/10.1093/biolinnean/blac080]

-
Hirase, S., Chambers, S., Hassell, K., Carew, M., Pettigrove, V., Soyano, K., Nagae, M., Iwasaki, W., 2017, Phylogeography of the yellowfin goby Acanthogobius flavimanus in native and non-native distributions, Mar.Biol., 164, 1-12.
[https://doi.org/10.1007/s00227-017-3137-6]

-
Hwang, J., Wong, C. K., 2005, The china coastal current as a driving force for transporting calanus sinicus (Copepoda: Calanoida) from its population centers to waters off taiwan and hong kong during the winter northeast monsoon period, J. Plankton Res., 27, 205-210.
[https://doi.org/10.1093/plankt/fbh162]

- Iwata, A., Jeon, S. R., 1987, First record of four gobiid fishes from Korea, 20, 1-12.
- Kim, I. S., 1987, A Taxonomic revision of the subfamily gobiidae (pisces, gobiinae) from Korea, 20, 529-542.
-
Kuno, Y., Takita, T., 1997, The growth, maturation and feeding habits of the gobiid fish Acanthogobius hasta distributed in ariake sound, Kyushu, Japan. 63, 242-248.
[https://doi.org/10.2331/fishsci.63.242]

- Lee, Y., 1992, A Taxonomic study of the genera Acanthogobius and Synechogobius (Pisces: Gobiidae) from Korea, 4, 1-25.
- Lee, Y., 2013, Changes in the list of species in Korean gobiid fishes, 24, 199-213.
- Moore, A., Giorgetti, A., Maclean, C., Grace, P., Wadhwa, S., Cooke, J., Prepared for the australian government department of the environment, water, heritage and the arts, commonwealth of Australia, 2008, Review of the impacts of gambusia, redfin perch, tench, roach, Yellowfin goby and Streaked goby in Australia, Australia.
-
Morgan, S. G., 1990, Impact of planktivorous fishes on dispersal, hatching, and morphology of estuarine crab larvae, 71, 1639-1652.
[https://doi.org/10.2307/1937574]

- Moyle, P. B., Cech, J. J., 2004, Fishes, 5th, Pearson Prentice Hall, Upper Saddle River, NJ.
- Nakabo, T., 2013, Fishes of Jjapan with pictorial keys to the species, Japan, 16-121.
- National Institute of Biological Resources (NIBR), National institute of biological resources, 2024, National list of Korea, Incheon, Korea.
-
Neilson, M. E., Wilson Jr, R. R., 2005, mtDNA singletons as evidence of a post-invasion genetic bottleneck in Yellowfin goby Acanthogobius flavimanus from San Francisco bay, California, Mar. Ecol. Prog. Ser., 296, 197-208.
[https://doi.org/10.3354/meps296197]

- Nelson, J. S., Grande, T. C., Wilson, M. V., 2016, Fishes of the world, 5th, John Wiley & Sons, Hoboken New Jersey, 326-333.
-
Raco, M., Jung, T., Horta Jung, M., Chi, N. M., Botella, L., Suzuki, N., 2023, Sequence and phylogenetic analysis of a novel alphaendornavirus, the first virus described from the oomycete plant pathogen phytophthora heveae, Arch. Virol., 168, 1-7.
[https://doi.org/10.1007/s00705-023-05786-7]

- Shin, H., Byun, D., Park, K., Lee, S., Kang, B., Lee, S., Choi, B., Lee, E., Korea hydrographic and oceanography agency, 2016, Research on creating a standard template for educational schematic current map, Seoul, Korea.
-
Song, N., Zhang, X. M., Sun, X. F., Yanagimoto, T., Gao, T. X., 2010, Population genetic structure and larval dispersal potential of Spottedtail goby Synechogobius ommaturus in the north‐west pacific, J. Fish Biol., 77, 388-402.
[https://doi.org/10.1111/j.1095-8649.2010.02694.x]

-
Song, N., Zhang, X., Gao, T., 2010, Genetic diversity and population structure of Spottedtail goby (Synechogobius ommaturus) based on AFLP analysis, Biochem. Syst. Ecol., 38, 1089-1095.
[https://doi.org/10.1016/j.bse.2010.10.007]

- Sun, Z., Li, Y., Song, C., Chen, J., Song, N., 2020, Comparative studies on morphology and genetics between Acanthogobius ommaturus and Acanthogobius flavimanus, JFC, 44, 1237-1248.
- Suzuki, N., Sakurai, N., Sugihara, T., 1989, Development of eggs, larvae and juveniles of the Oriental goby Acanthogobius flavimanus reared in the laboratory, 36, 277-289.
-
Thuy, T. T., My, N. H., Hau, T. D., 2022, First faunal record of larval and juvenile gobies (Actinopterygii: Gobiiformes) in the ka long estuary, northern vietnam, 44, 61-72.
[https://doi.org/10.15625/2615-9023/16120]

-
Zhang, D., Kan, X., Huss, S. E., Jiang, L., Chen, L., Hu, Y., 2018, Using phylogenetic analysis to investigate eukaryotic gene origin, 138, e56684.
[https://doi.org/10.3791/56684-v]

-
Zhang, D., Gao, F., Jakovlić, I., Zou, H., Zhang, J., Li, W. X., Wang, G. T., 2020, PhyloSuite: An integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies, 20(1), 348-355.
[https://doi.org/10.1111/1755-0998.13096]

Department of Marine Biological Resource and Aquaculture, Kunsan National Universitychivalry96@naver.com
Department of Marine Biological Resource and Aquaculture, Kunsan National Universitychoi@kunsan.ac.kr
Department of Marine Biological Resource and Aquaculture, Kunsan National Universityleekiy@kunsan.ac.kr