CRISPR/Cas9을 이용한 ZmWUS1 유전자 promoter 교정 및 옥수수 형질전환 개선
Ⓒ The Korean Environmental Sciences Society. All rights reserved.
This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
Along with wheat and rice, maize has the highest yield and cultivation area in the world. With the development of gene editing technology, methods are being proposed to easily and quickly breed new varieties by applying new technologies to cultivating varieties using traditional breeding methods. However, transformation technology to introduce CRISPR/Cas9 constructs for gene editing into maize (Zea mays) has already been developed and used in the West, but in Korea, there are still no domestic research institutes or universities that are constructing and carrying out transformation using Hi-II A/B cultivars for transformation. This study was performed to establish maize transformation in Korea using Hi-II A/B cultivar. We cloned guide RNA constructs targeting the promoter region of the maize WUSCHEL1 (ZmWUS1) gene, a homeobox transcription factor deeply involved in shoot apical meristem (SAM) maintenance and regulating crop productivity. The confirmed CRISPR/Cas9 construct containing the guide RNA was successfully introduced into maize via Agrobacterium transformation method, and sequencing analysis confirmed that gene editing occurred in the target region. One of the ZmWUS1 promoter mutations by target edition displays abnormal ear development. This study is expected to contribute greatly to the breeding of new maize varieties by disseminating maize transformation technology domestically and revitalizing molecular biology and molecular genetics research on maize.
Keywords:
CRISPR/Cas9, Genome edition, Hi-II A/B, Maize transformation, ZmWUS1 promoter1. 서 론
유전자교정 기술은 외래 유전자를 남기지 않으면서도 자연 돌연변이와 유사한 안정성을 제공하는 기술이다. 이는 식량 수급 안정화, 기후변화 대응 작물 개발, 작물의 산업적 활용 확대 등 다양한 작물 개량 분야에서 획기적인 돌파구로 주목받고 있다. 특히 세계적 주요 곡물로서 식량 안보와 산업적 이용 가치가 높은 단자엽 모델 작물 옥수수(Zea mays L.)의 경우 유전자교정(Brooks et al., 2014; Doudna and Charpentier, 2014; Belhaj et al., 2015)을 통한 작물 개발의 성공 여부는 재분화 및 형질전환의 가능성 유무에 따라 결정되어 왔다. 미국에서는 2000년대 초반부터 옥수수 형질전환 기술이 활발히 연구되고 있다. 특히 Dupont-Pioneer와 같은 대형 종자회사는 자체 연구소를 설치하여 꾸준히 기술을 향상시키고 있고(Che et al., 2018; Lowe et al., 2018), 또한 유럽을 비롯한 중국 등 많은 나라들이 이미 옥수수 형질전환을 상용 기술로 사용하고 있다.
현재 연구자들은 A188과 B73을 교배해 형질전환에 적합한 Hi-II A와 Hi-II B 계통을 선발한 후, 이 두 계통을 교배한 F2 개체에서 어린 배아를 적출하여 옥수수 형질전환에 널리 활용하고 있다. 그러나 Hi-II A와 Hi-II B 계통은 생장 과정에서 다른 품종에 비해 식물체 활력이 떨어지므로, 숙련된 재배 경험이 없으면 자가수분이나 교배 수분을 위한 개화기 일치와 실효 수분을 달성하기 어렵다(González et al., 2012). 그러나 품종 육종에 직접 이용될 수 있는 일반 계통에서는 Agrobacterium 감염에 대한 저항성으로 인해 형질전환 효율이 현저히 감소하므로 실질적인 재배 계통에 대한 형질전환에 한계가 있다(Zhao et al., 2002; Huang and Wei, 2005; Wang et al., 2007; Vega et al., 2008). 최근에는 생장점의 활성도에 관여하는 두 유전자(ZmBBM and ZmWUS2)를 함께 과발현시켜 일반 inbred 계통에서도 형질전환의 효율이 개선된다고 보고하였다(Lowe et al., 2016).
국내의 옥수수 재분화 연구에서는 농진청과 같은 큰 연구기관에서 HI II 계통을 포함한 다양한 계통을 이용하여, 최적의 순계 계통 선발과 형질전환법 개발, 제초제 저항성 옥수수 생산을 목표로 한 연구들이 보고되었다(Cho et al., 2005; Kim et al., 2014; Hong et al., 2017; Hong et al., 2019). 하지만 옥수수 형질전환 기술이 국내 소규모 연구실에서 실행될 수 있는 보편화 플랫폼 구축이 마련되지 않았고, 또한 CRISPR/Cas9 유전자교정 기술을 접목한 옥수수 형질전환 기술은 국내에서는 전무한 실정이다.
본 연구에서는 옥수수 형질전환 기술의 국내 보편화 플랫폼 구축을 위해 일반 소규모 실험실에서 주로 사용되는 조직배양 생장상(Growth chamber)을 이용하여 실험을 진행하여, 소규모 실험실에 최적화된 수정된 옥수수 형질전환 프로토콜(Protocol)을 제안하고자 수행되었다. 이를 위해 우리는 생장점 발달에 관여하는 ZmWUS1 유전자의 프로모터(Promoter)를 표적으로 CRISPR/Cas9 기반 유전자 편집 기술을 적용하여 성공적인 유전자편집 형질전환을 확인하였다.
2. 재료 및 방법
2.1. 실험재료 및 식물재배
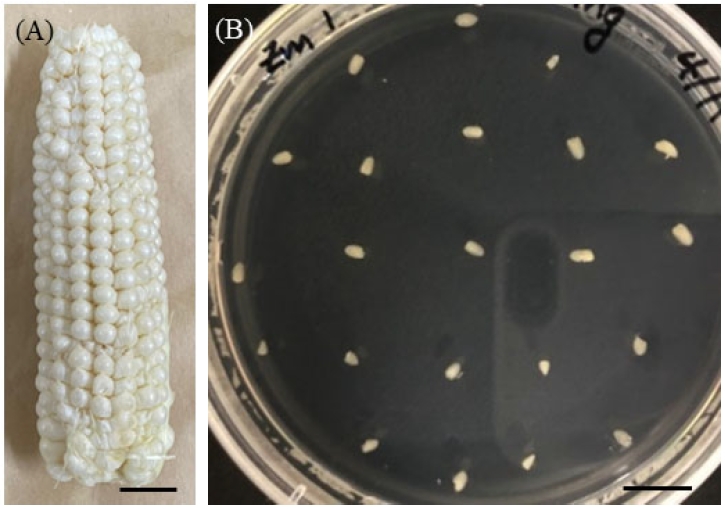
옥수수 형질전환용 계통 Hi-II A와 Hi-II B는 밀양 캠퍼스 첨단 온실과 부산대학교 부속 농장에서 각각 재배하였다. 먼저 Hi-II A와 Hi-II B를 개별적으로 재배하고 두 계통을 교배하여 F1을 생산한 후, 다음 시즌에 F1을 파종하여 자가 수분 된 F2 개체의 어린 이삭에 있는 배아를 이용하였다. 노지에서는 재배 기간동안 평균 약 25°C, 강수량 255 mm의 환경에서 재배하였고 첨단 온실에서는 자동 온도 조절 시스템을 이용하여 26 ± 2°C에서 16시간 명기, 8시간 암기의 생육환경을 설정하여 재배하였다. 첨단 온실에서 포트 재배 시 파종 후 1달 뒤와 수술이 올라올 때 지름 32 cm 화분 기준 줄기를 피해 화분의 끝부분에 10 g의 질소비료(슈퍼 NK; 천지 바이오)를 땅에 파묻어 시비하였다. Hi-II A/B F2의 어린 이삭은 수분 후 12 ± 2일 된 미성숙 접합체 배아(Immature zygotic embryo)가 약 1.5-2 mm일 때 수확하였다(Fig. 1A). 수분 후 10일부터, 배아의 크기를 결정하는 온도, 강수량, 일조량과 같은 환경 조건을 고려하여, 이삭을 수확하지 않고 옥수수 수염의 husk leaf을 열어 배아 크기를 매일 확인해 형질전환 시기를 확인하였다. 어린 이삭은 수확일에 바로 형질전환에 사용되거나 그렇지 못할 경우 껍질 잎(Husk leaf)에 싸인 채로 교배대(Pollination bag)에 넣어 4°C 냉장고에 1-2일 보관하여 형질전환에 사용하였다.
2.2. Transgene construction
옥수수의 화서 발달 관련 ZmWUSCHEL1 (ZmWUS1, GRMZM2G047448) 유전자의 편집을 위해 https://www.maizegdb.org/ 사이트로부터 유전자 정보를 확인하고(Gene: Zm00001eb067310, hb67-Homeobox transcription factor 67), CRISPR direct software (https://crispr.dbcls.jp/)를 이용하여 Single guide RNA (sgRNA)를 제작하였다. sgRNA는 CRISPR/Cas9 효소가 인식하는 PAM (Proto spacer adjacent motif) 영역인 5’-NGG-3’ 서열과 20bp의 표적 염기 서열로 이루어져 있다. sgRNA는 off-target이 없는 서열과 40-60%의 GC 함량을 가진 것을 선택하였다. 프로모터 영역은 유전자의 발현을 조절하는 중요한 영역이므로 8개의 영역을 타겟으로 유전자 편집을 유도해 효과적으로 발현 수준을 조절하고자 하였다. 본 연구에서는 sgRNA가 벼 U3 promoter로 발현되고 옥수수에 codon optimization 된 Cas9 효소가 옥수수 Ubiquitin promoter로 발현되는 벡터(Vector)를 사용하였다. 재조합된 식물 발현 벡터는 Agrobacterium tumefaciens EHA105과 LBA4404 균주에 각각 형질전환하였다. 형질전환체의 선발은 제초제는 비알라포스(Bialaphos)를 이용하였다. gRNA design을 위한 프라이머 리스트는 Table 1에 작성하였다.
2.3. 형질전환체 제작과 유전자 도입 확인
확인된 클론은 Agrobacteria EHA105과 LBA4404 strain으로 옮겨졌다. 형질전환된 Agrobacteria를 rifampicin 50 mg/L과 spectinomycin 50 mg/L이 함유된 YEP 액체 배지에 28°C에서 2일 배양한 후 배양액을 rifampicin 50 mg/L과 spectinomycin 50 mg/L이 함유된 YEP 고체배지에 100 uL을 도말하여 다시 28°C에서 2일 배양하였다. 배양된 Agrobacteria clone을 긁어 infection 배지 5 mL에 넣고 1시간 동안 28°C로 유지되는 교반기(Shaker)에서 배양해 현탁액이 완전히 풀어지도록 하였다. 준비된 현탁액은 큐벳을 이용해 분광 광도계(Spectrophotometer)로 OD600 값을 측정하였고 현탁액의 최종 농도가 OD600 = 0.5이 되도록 acetosyringone stock 100 mM/mL이 포함된 infection 배지로 희석하여 형질전환에 사용하였다.
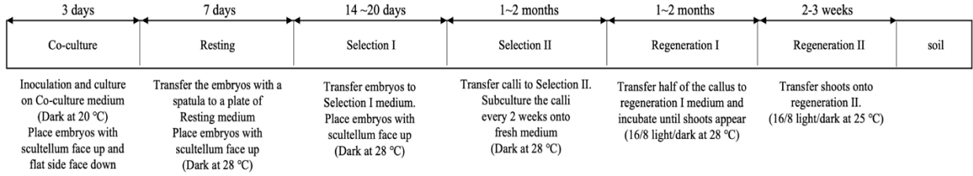
옥수수 형질전환에 사용된 조직은 수분 된 지 약 12일 지난 어린 배아로 크기가 약 1.5-2 mm가 적당하였다(Mohanty et al., 2009). 배아 크기가 적당해진 Hi-II A/B F2 어린 이삭의 겉껍질을 제거하고 멸균된 약수저(Stainless steel spatula)를 이삭 중앙에 꽂아 손잡이로 사용하였다. 5 L 플라스크에 2 L의 락스, 2 L의 멸균된 3차 증류수, 그리고 80 uL Tween 20을 섞어 완전히 잠기도록 어린 이삭을 넣은 후 30분 동안 소독한다. 30분 동안 손잡이를 가볍게 휘저어주었다. 멸균된 3차 증류수로 다섯 번 이상 완전히 락스를 씻어낸 후, 발달 중에 있는 어린 배아를 적출하였다. 여러 개의 이삭이 있는 경우 이삭이 가득 담기게 멸균된 3차 증류수를 채워 담겨주도록 하였다. 이삭에 꽂아 둔 손잡이가 위로 가도록 이삭을 잡고 위에서 아래로 1줄씩 낱알의 2/3이 남도록 횡축으로 윗면을 잘라낸다. 멸균된 스패츌러로 낱알의 오른쪽 옆을 끝까지 찔러 전분 부분을 들어내고 가장 아랫부분에 있는 배아를 추출했다. 이때 아랫부분부터 약수저로 찔러 배아가 상처를 입거나 부서지지 않도록 주의한다. 10분 동안 1 mL의 infection 배지가 담겨 있는 2.0 mL의 micro-centrifuge tube에 추출된 배아를 모은다. 10분 후 infection 배지를 피펫(Pipette)으로 제거한 뒤 3차례 1 mL의 새로운 acetosyringone stock 100 mM/mL이 포함된 infection 배지로 씻어내고 1.5 mL의 Agrobacterium 현탁액을 첨가해 5분 동안 천천히 튜브를 위아래로 흔들어주며 감염시킨다. 5분 뒤 현탁액을 포함한 배아들을 co-culture 배지에 모두 쏟아붓는다. 배지에 남은 현탁액들을 모두 제거하고 배아의 배반이 위로 향하고 배아 쪽이 배지에 닿도록 배치한 뒤 30분 동안 배지를 말려 뚜껑을 닫고 미세기공 테이프(Micropore tape)로 동봉하여 20°C 암 상태의 챔버에서 3일간 배양하였다(Fig. 1).
공동배양이 끝난 배아들은 resting 배지로 배반이 위로 향하게 옮겨져 28°C에 암 상태의 식물 배양 챔버에서 7일 동안 배양하였다(Fig. 2). 선발배지 I 부터는 1.5 mg/mL의 비알라포스(Bialaphos) 제초제를 첨가하여 28°C 암 상태에서 14일-20일 동안 배양하였다. 캘러스가 분화되기 시작하는 배아는 비알라포스 3 mg/mL가 첨가된 선발배지 II로 옮겨져 28°C에 암 상태에서 배양하였다. 캘러스 분화로 직경이 1.5 cm가 넘는 캘러스는 반을 떼어내어 재분화 배지 I로 옮겨 green spot과 신초를 유도하였다. 재분화 배지로 옮긴 캘러스부터는 28°C에서 16시간 명기, 8시간 암기를 가지는 챔버에서 배양하였다. 신초가 1.5 cm 이상 자라면 재분화 배지 II로 옮겨 25°C에서 16시간 명기, 8시간 암기를 가지는 챔버에서 뿌리를 유도하였다. 뿌리까지 성공적으로 재분화된 형질전환체는 흙으로 옮겨 챔버에서 순화과정을 거친 후 직경 32 cm 화분에 정식되어 부산대학교 첨단 온실에서 재배하였다.
형질전환체에서 재조합 벡터의 도입을 확인하기 위해 재분화 된 개체의 잎의 genomic DNA를 주형으로 Phosphinothricin N-acetyltransferase (PAT) 유전자 특이 증폭 프라이머를 이용하여 PCR 검정을 수행하였다. 사용한 프라이머의 서열은 PAT-forward 5'-TGAAGTCCAGCTGCCAGAAA-3', PAT-reverse 5'- ACATCGAGACAAGC ACGGTC-3'으로 제작되어 분석에 사용하였다.
2.4. 형질전환체 염기 서열 분석
PAT 유전자 증폭 프라이머 검정 결과로 선발된 형질전환 돌연변이체의 염기 서열을 알아보기 위해 옥수수 ZmWUS1 유전자 프로모터 영역의 2 kbp에 걸쳐 총 3개의 목표 서열 특이 프라이머 세트를 제작하였다. 각각 WUS1-geno-F1 5'-GGCTTTACGTCGATCTCAGG-3', WUS1-geno-R1 5'-CGGTACCGCTATGTATGTGTG-3', WUS1-geno-F2 5'-GAAACAGGGAGTTGCTCGAT-3', WUS1-geno-R2 5'-AGAGAGAGCGCATGCAGAGT-3', WUS1-geno-F3 5'-GAGCGAGCAATGATGCACTA-3', WUS1-geno-R3 5'-CCTTGAGGATCCTGA TCTGCT으로 제작되어 PCR 검정에 사용하였다. Hi- II A/B F2과 각각의 형질전환 개체들은 3개의 프라이머 세트로 각각 1,144 bp, 614 bp, 970 bp로 증폭하였다. PCR 산물은 Exo-CIP Rapid PCR Cleanup Kit (New England Biolabs-NEB, Ipswich, MA, USA)를 사용하여 정제한 후 (주)바이오니아(www.bioneer.co.kr)로 의뢰하여 염기 서열을 분석하였다.
2.2 통계분석
형질전환은 Agrobacteria EHA105과 LBA4404 strain를 가지고 총 4 반복으로 수행하였다. 접종한 배아의 수, 비알라포스(Bialaphos)에 선발된 캘러스의 수, 재분화 효율 및 유전자 편집 효율 결과의 값은 평균(Mean) ± 표준오차(Standard error, SE)로 나타내었다. 통계분석은 SAS 프로그램(SAS9.4, SAS Institute Inc., USA)을 이용하여 일원분산분석(One-way ANOVA)으로 처리 간 평균의 차이를 검증하였다. 사후검정으로는 Duncan’s multiple range test를 사용하였으며, 유의수준은 5%로 설정하여 처리 평균 간 통계적으로 유의한 차이가 있는지 확인하였다.
3. 결과 및 고찰
3.1. 옥수수 ZmWUS1 유전자 동정과 CRISPR/Cas9 벡터 구축
애기장대에서 줄기세포의 항상성은 WUSCHEL (WUS) 과 CLAVATA 사이의 음성 피드백에 의해 제어되는 것으로 알려져 있다(Fletcher, 2018; Schoof et al., 2000). 벼 유전체 상에는 단일 WUS 상동 유전자(Ortholog), OsWUS/OsTAB1이 존재하는 반면, 옥수수 유전체에는 2개의 WUS 이원체(Paralog)인 ZmWUS1과 ZmWUS2가 보고되어져 있다(Nardmann, 2006). 옥수수에서 ZmWUS1은 화서 발달과 화서 꽃차례에 밀접한 관련이 있는 것으로 예측되며 더 자세히 알아보기 위해 먼저 AtWUS와 64% 동일성을 갖는 320개 아미노산의 단백질을 암호화하는 옥수수 동원체 중 하나인 ZmWUS1 (hb67)을 동정하였다(Chen et al., 2023). ZmWUS1의 프로모터 영역에 유전자 편집을 하여 표현형을 관찰하고자 하였는데, 프로모터 영역은 유전자의 발현을 조절하는 중요한 부분으로, 여기에 가해지는 변화는 해당 유전자의 발현 수준에 직접적인 영향을 주기 때문이다. 특히, 화서 발달 단계나 화서 구조의 형성에 영향을 미치는지 알아보고자 다음과 같이 벡터를 구축하였다. 유전자 편집 돌연변이를 위한 표적은 골든 게이트 클로닝(Golden gate cloning) 방식을 사용하여 총 8개의 목표 서열에 동시에 편집할 수 있는 벡터 construct를 구축하였다. 본 연구에서는 하나의 목표 서열을 편집하는 것보다 8개의 서열을 편집하여 특정 서열이 효과적으로 수정되지 않을 경우 다른 서열을 선택하여 높은 효율로 유전자 편집이 되도록 유도하였다.
다수의 표적 서열을 각각 조립하려면 복잡한 제한효소와 DNA 연결효소를 이용한 유전자 재조합 작업이 필요할 것이다. 이는 실험의 복잡성을 증가시키고 실험자가 여러 단계의 조립을 수동으로 벡터를 재조합해야 함을 의미한다. 이러한 어려움을 해결하기 위해 본 연구에서는 다중 표적(Multiple targeting) CRISPR/Cas9 construct를 구축할 수 있는 내인성 t-RNA 처리 시스템(Endogenous t-RNA processing system)을 활용하였다(Xie et al., 2015; Qi et al., 2016; Xie and Yang, 2019). 8개의 목표 염기 서열에 대한 각각의 tRNA-sgRNA-scaffold fragment는 pGTR 벡터를 주형으로 합성하였다. 첫 번째와 마지막 조각은 각각 entry 벡터와 상보적인 접착성 말단(Sticky end)이 설계된 BsaI site와 FokI site를 가진 "L5AD5-F"와 "L3AD5-R" 프라이머를 사용하여 증폭하였다. 두 번째 조각은 BsaI site, 첫 번째 타겟 spacer의 3’ 염기 서열 부분의 반, 그리고 scaffold 서열의 일부분을 가지는 정방향 프라이머와 BsaI site, 두 번째 타겟 spacer의 5’ 염기 서열 부분의 반, 그리고 tRNA 서열의 일부분을 가지는 역방향 프라이머를 사용하여 제작하였고, 이후의 7개의 조각들도 동일한 방식으로 제작하였다. 제작된 총 9개의 조각은 골든 게이트 클로닝 방식을 사용하여 하나로 조립되어 "S5AD5-F"와 "S3AD5-R" 프라이머로 한 번 더 증폭된 후, pOS-sgRNA entry 벡터의 U3 프로모터 뒤에 BsaI type IIS 효소를 사용하는 골든 게이트 클로닝 방식을 이용하여 삽입하였다. pU3-[tRNA -sgRNA1-Scaffold] x 8 fragment는 Gateway 클로닝 방식으로 LR clonase II를 사용하여 Cas9 유전자와 2X CaMV 35S 프로모터로 조절되는 제초제 Phosphinothricin 분해 유전자(PAT)가 포함된 최종 벡터(Destination vector, pMCG1005)에 성공적으로 클로닝 하였다(Fig. 3).
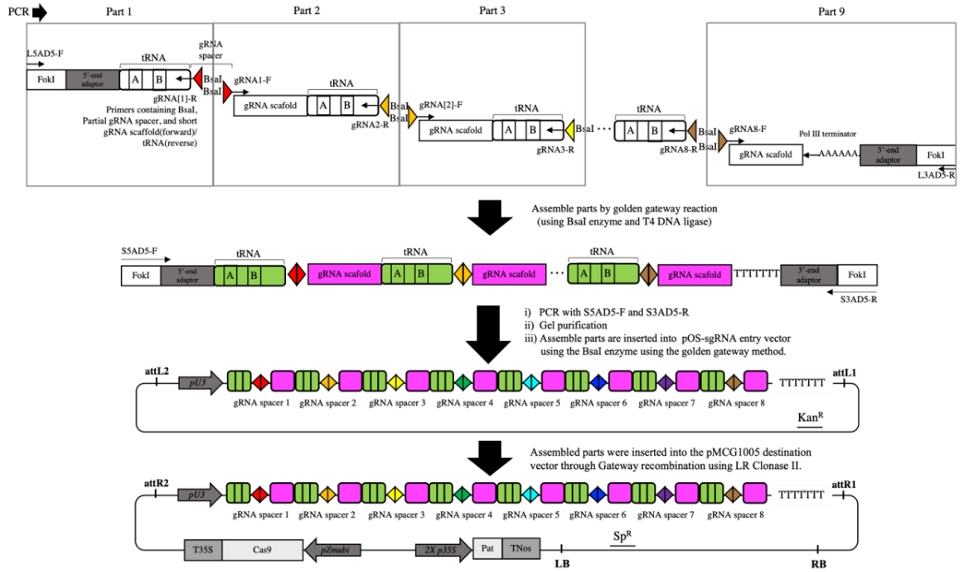
Schematic overview of multiple target CRISPR/Cas9 genome editing in maize utilizing the endogenous t-RNA processing system cloning process. The fragment that has been ligated into the entry vector using golden gateway system and is recombined into the expression vector through LR reaction. pU3, rice U3 promoter driven pOS-sgRNA; TTTTTTTT, Polymerase III terminator; Cas9, maize codon-optimized Cas9; pZMubi, maize ubiquitin promoter; T35S, CaMV 35S terminator; 2X p35S, CaMV 35S promoter; TNos, Nos terminator; Pat, Basta resistance gene; LB, left border; RB, right border; attL1, attL2, attR1, attR2, gateway left and right border sequence.
3.2. 옥수수 형질전환 배지조성 개선
단자엽 식물은 Agrobacteria의 자연 숙주가 아니기 때문에 옥수수에서 Agrobacteria 매개 형질전환을 통한 외래 유전자 도입은 미성숙된 배아, 성숙된 배아, 중배축, 약, 신초, 잎, 미성숙 수술과 이삭에서 시도되었다(Torne et al., 1980; Ting et al., 1981; Songstad et al., 1992; Sidorov et al., 2006; Ahmadabadi et al., 2007; ZHAO et al., 2008). 이 중 가장 높은 효율로 보편적으로 사용되고 있는 방법은 미성숙 배를 통한 형질전환으로 알려져 있다(Kang et al., 2022). 미성숙 배를 사용해 얻을 수 있는 캘러스는 두 가지 유형이 알려져 있는데, type I 캘러스는 치밀하고 단단하며 재분화가 잘 안되지만, type II 캘러스는 부드럽고 단단하지 않아 부서지기 쉬운 구조로 이루어져 있고 빠르게 재분화되는 특징을 가진다. 효율적인 옥수수 형질전환 시스템을 위해서는 type II 캘러스를 생산하는 식물 계통을 사용하는 것이 효율적이라는 보고가 되어져 있다(Armstrong and Green, 1985). 특히 A188과 B73의 교배로부터 유래되어진 Hi-II A와 B계통은 미성숙 배에서 전형적인 type II 캘러스를 생산할 수 있게 하고 다른 계통을 사용해 형질전환을 했을 때 보다 더 높은 재분화능을 보이는 것으로 보고되어 옥수수 형질전환 연구에서 중요한 유전자원으로 알려져 왔다(Zhao et al., 2002). Hi-II A와 B계통을 각각 이용한 옥수수 형질전환법은 기존에 많이 알려져 있지만 형질전환율이 아주 낮거나 보편적으로 캘러스가 형성되다가 green spot을 띄는 캘러스 상태에서 괴사하거나 캘러스가 분화되다 멈추고 백화현상이 나타나는 등 기존의 방식(Frame et al., 2002)에는 재분화를 시키는 과정에 문제가 있었다(Fig. 4A, B).

Modification of medium composition resulted in healthy and embryonic callus. Before the medium composition was modified, callus differentiated and progressed to chlorosis, non-embryogenic callus, or necrosis (A, B). Increasing the concentration of auxin and cytokinin in the medium composition enabled successful redifferentiation of callus and the appearance of green spots (C, D). Scale bar, 0.3 cm.
한편 옥수수와 같은 단자엽 식물인 벼에서는 옥수수와 다르게 보편적으로 사용할 수 있는 Agrobacteria 매개 형질전환 방법이 알려져 있어 옥수수에서도 다양한 형질전환법에 대한 시도가 보고되고 있다(Hiei et al., 1994; Sahoo et al., 2011; Sahoo and Tuteja, 2012). 효율적인 형질전환에 영향을 미치는 요소에는 Agrobacteria 균주의 선택과 Agrobacteria 접종 효율이 높고 캘러스 분화에 유리한 식물 재료의 최적화, 도입 벡터의 선택과 조직 배양 배지의 최적화 등이 고려되어야 한다(Gelvin, 2003). 본 연구에서는 식물 재료로 캘러스 분화율이 높다고 알려진 Hi-H II A와 B계통을 사용하였기 때문에 형질전환을 위한 계통 선택의 잘못보다는 조직 배양 배지의 생장조절제의 함량에 문제가 있다고 판단하였고, 기존의 프로토콜(Frame et al., 2002)을 조금씩 수정하여 형질전환을 시도하였다.
식물 형질전환에 사용되는 전형성능을 가진 캘러스를 유도하려면 배양 배지에 2,4-D 또는 유사한 옥신 생장조절제 그리고 사이토키닌이 존재해야 한다. 조직배양 배지에 사용될 수 있는 사이토키닌의 종류는 kinetin, Thidiazuron, zeatin, 6-Benzylaminopurine hydrochloride (6-BAP) 등이 있다(Werner Tomáš, 2001). 벼의 형질전환 과정에서 재분화를 위한 사이토키닌은 주로 kinetin이 사용되는 것으로 알려져 있고, 옥수수에서는 황산구리(CuSO4)와 함께 6-BAP가 사용되는 방법이 보고되어져 있다(Cho et al., 2014; Wu et al., 2014). 본 연구에서는 벼의 형질전환법에서 착안하여 2,4-D와 kinetin의 혼용이 황산구리와 6-BAP를 함께 시용하였을 때처럼 캘러스 분화율을 증가시키고 신초를 유도할 수 있는지 알아보고자 하였다.
동일한 단자엽 식물인 벼의 Agrobacterium 매개 형질전환법에서 사용되는 2 mg/mL의 2,4-D가 함유된 induction과 선발배지는 캘러스 분화가 빠르고 확실하게 일어나는 것에서 착안하여 Selection I의 배지조성에서 첨가되는 2,4-D (1 mg/mL stock)의 양을 1.5 mg/mL에서 2 mg/mL로 옥신의 농도를 높여주었다(Muppala et al., 2020). Selection II 배지에서는 2,4-D (1 mg/mL stock)의 과도한 축적을 막기 위해 1.5 mg/mL에서 1 mg/mL로 옥신의 농도를 다시 줄여 주었고, 기존 프로토콜에는 첨가되지 않았던 사이토키닌 kinetin (1 mg/mL stock)을 2 mg/mL 첨가하였다. 그리고, Regeneration I에서도 기존에 없었던 사이토키닌 kinetin (1 mg/mL stock)을 3 mg/mL 첨가하였다(Table 2). 생장조절제의 조성은 벼의 형질전환 체계법을 참고하였지만, 옥수수 배아를 사용한 형질전환에서 캘러스 분화를 돕기 위해 L-cystein과 silver nitrate (AgNO3)을 함께 첨가하여 배지조성을 개선하였다(Carvalho et al., 1997; Yassitepe et al., 2021).
3.3. 옥수수 형질전환체 생산
Agrobacteria 균주는 형질전환 효율에 중요한 영향을 미치는 요인 중 하나이다. 기존 연구에 따르면, LBA4404는 약 ~5%의 형질전환 효율을 보이며, 옥수수 형질전환에 보편적으로 사용되던 EHA105와 큰 차이가 없는 것으로 보고되었다(Huang and Wei, 2005; Ishida et al., 2007). 본 연구에서는 수정된 배지 조성을 사용하여 형질전환을 4회 반복 수행한 결과, Agrobacterium 균주 EHA105를 사용하여 형질전환 하였을 때 평균 71.0 ± 7.2개의 미성숙 배아 중 4.25 ± 1.3개의 재분화된 비알라포스 저항성 캘러스를 확보하였으며, 재분화 효율은 5.99%로 나타났다. 반면, LBA4404를 사용했을 때는 평균 53.5 ± 12.77개의 미성숙 배아 중 4.0 ± 1.8개의 비알라포스 저항성 캘러스를 확인하였으며, 형질전환율은 7.48%로 나타났다. 이 결과는 국내 옥수수 형질전환 기술의 형질전환율(약 2%)보다 높고, Hi II A/B F1의 미성숙 배아를 사용하여 형질전환을 수행한 국제 연구에서 보고된 형질전환율(5.5-12%) 범주에 포함된다(Cho et al., 2005; Lee and Zhang, 2016; Hong et al., 2019). 이러한 결과는 초기 캘러스 선발 과정에서 옥신 농도를 1.5 mg/mL에서 2 mg/mL로 증가시키고, 사이토키닌인 kinetin 농도를 2-3 mg/mL로 조정한 것이 캘러스 재분화율과 형질전환율 향상에 기여했음을 시사한다. 특히, 사이토키닌을 함유하지 않는 배지에서 형질전환을 하였을 때는 재분화 캘러스를 얻지 못했기 때문에 조직 배양에서 옥신뿐만 아니라 사이토키닌 농도가 캘러스 재분화 및 신초, 뿌리 분화에 필수적인 요인임을 확인하였다(Fig. 4C, D and Fig. 6).

PCR confirmation of T0 transformants through PAT gene amplification. The expected size of the PCR product is 422 bp. Size marker, 100 bp DNA ladder.
한편, EHA105로부터 얻어진 저항성 캘러스는 Type II 캘러스는 분화되었지만 모두 신초 분화가 일어나지 않고 그 상태로 고사해버린 반면에, LBA4404를 사용했을 때는 6개의 ZmWUS1 형질전환체(Transgenic events)를 확보할 수 있었고, 이에 대한 유전자 편집 효율은 2.9 ± 1.4%로 나타났다(Table 3). 이러한 결과는 HI-II A/B F1 계통의 선택, pMCG1005 벡터에 포함된 유전자 구성 요소, LBA4404 균주, 그리고 옥신과 사이토키닌 비율의 최적화가 형질전환 효율 향상 및 CRISPR/Cas9 시스템 도입 성공에 기여했음을 보여준다(Ishida et al., 1996; Shrawat and Lörz, 2006; Loyola-Vargas and Ochoa-Alejo, 2016; Horstman et al., 2017).
3.4. Promoter ZmWUS1 region의 유전자 편집 확인과 표현형 검정
도입유전자가 삽입된 T0 개체들로부터 유전자 편집 여부는 ZmWUS1 유전자의 프로모터 영역 2 kbp를 증폭하는 프라이머를 제작 이용하여 PCR을 수행하고 프로모터 영역에서 유전자 편집이 일어난 것을 확인하였다. 재조합 벡터가 도입된 것으로 확인된 6개의 형질전환체 중 3개체에서 유전자의 프로모터 영역에서 돌연변이를 확인하였다. 첫 번째 변이체 ZmWUS1CR-mutant 1은 3번째 4번째 타겟 사이 서열의 1 bp (A) 결실, 두 번째 변이체 ZmWUS1CR-mutant 2는 5번째 유전자 목표 서열 바로 앞에 1 bp (A) 결실, 세 번째 변이체 ZmWUS1CR-mutant 3은 7번째 유전자 gRNA 서열 앞의 2 bp (CC) 결실과 7번째 타겟의 7번째 염기 A가 C로 치환된 것으로 확인하여 총 3 events를 선발하였다. 3개의 돌연변이체 모두 단일 유전자의 두 대립유전자 중 하나의 대립유전자에서 발생하는 돌연변이를 얻었다(Fig. 7). 3개의 서로 다른 events 돌연변이체 각각의 T1 세대는 대체로 생육이 저해되는 것을 제외한 특별한 표현형은 찾아보기 힘들었다. 하지만 ZmWUS1CR-mutant 3의 20개의 T1 개체 중 하나의 돌연변이체가 비정상적으로 많은 ears를 하나의 husk leaf 안에 자라나는 표현형을 보였다(Fig. 8). 하지만 자가수분을 통한 동형접합 자식 세대를 얻을 수 없어, 순계(B73)에 교배하여 T1 세대의 종자를 수확하였다.
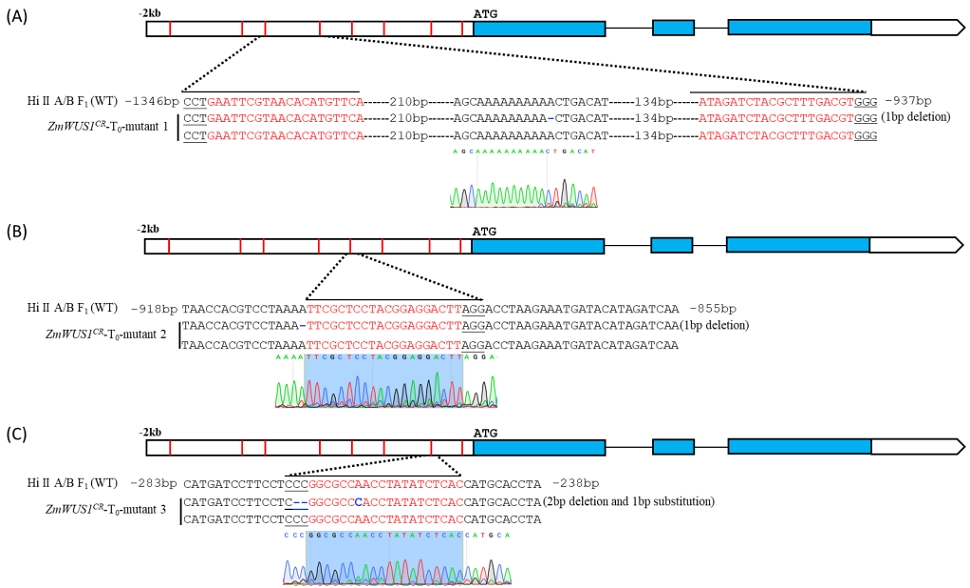
Schematic diagram of the eight target sites of the ZmWUS1 gene promoter edition and edition confirmation via Sanger sequencing of T0 plants. The red lines within the white boxes represent the edition target sites and blue boxes indicate exons of ZmWUS1. Sequences in red indicate the target region. 1 bp deletion in ZmWUS1CR-T0-mutant 1 was identified between the third and fourth target region (A). 1 bp deletion in ZmWUS1CR-T0-mutant 2 was identified just before the fifth target region (B). 2 bp deletion and 1bp substitution in ZmWUS1CR-T0-mutant 3 was identified in the seventh target region (C).
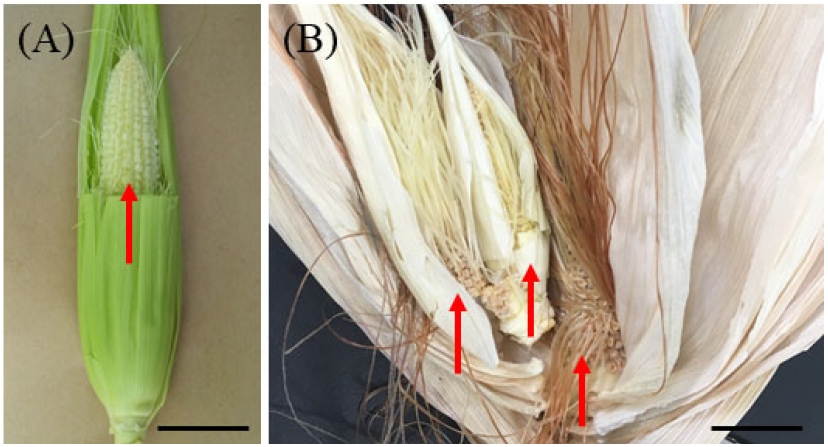
ZmWUS1CR-mutant 3 exhibits multiple ear development in a single shell. One ear of the wild type in a single shell (A) was compared with multiple ears of ZmWUS1CR-mutant 3 (B). Arrows indicate each ear. Scale bar, 2 cm.
본 연구는 옥수수 생장점의 크기와 균형적인 분열조직의 증식을 조절하는 CLV-WUS 신호전달 기작의 구성 요소 ZmWUS1 유전자 promoter를 표적으로 CRISPR/Cas 유전자교정을 형질전환을 진행하였다. CLV-WUS으로 대표되는 생장점 조절에서 ZmWUS1은 최종 신호전달 종착지 전사인자이다. 이는 작물의 생장과 발달에 필수적인 여러 줄기세포 중에서도 핵심이 되는 영양 생장기의 경단줄기세포(Shoot apical meristem, SAM)를 조절하고, 개화기 생장점(Inflorescence meristem, IM)의 크기 조절을 통한 작물의 생산성에 직결되는 열매의 크기와 수량을 결정하는 인자이다. 최근 연구에서는 옥수수의 Barren inflorescence3 (Bif3) 돌연변이형에서 ZmWUS1 유전자의 프로모터 서열 중 ZmWUS1-B의 중복 사본이 생기는 변이가 확인되었다. 이 변이로 인해 ZmWUS1의 발현이 증가하며, 어린 이삭의 발달 패턴에 영향을 미치는 것으로 보고되고 있다. 특히, 이 변이는 ZmWUS1CR-mutant 3과 유사한 표현형을 나타내며, 이는 ZmWUS1 유전자의 프로모터 영역의 제7 표적 gRNA 서열 앞의 2 bp CC 결실과 제7 표적의 7번째 염기 A가 C로 치환된 것이 ZmWUS1 유전자의 발현과 ear 발달에 관여한 것으로 보인다. 이처럼 ZmWUS1과 promoter region을 표적으로 유전자 편집을 활용한 다양한 ZmWUS1 발현 대립형질들은 화서 발달의 진화가 어떻게 옥수수 야생형 teosinte과 같은 alternate 대생 화서(꽃차례)에서 현대의 maize의 whorl type의 윤생 화서로 진화되었는지는 풀리지 않은 식물 발달과 진화의 큰 난제 중에 하나의 실마리를 제공할 것으로 기대된다.
4. 결 론
본 연구는 옥수수 생장점의 크기와 균형적인 분열조직의 증식을 조절하는 CLV-WUS 신호전달 기작의 구성 요소인 ZmWUS1 유전자의 프로모터를 표적으로 한 CRISPR/Cas9 기반의 유전자 편집 시스템을 이용하여 소규모 실험실에 최적화된 옥수수 형질전환 protocol 구축을 위하여 수행되었다. 옥수수 ZmWUS1 유전자의 프로모터 영역에 총 8개의 목표 서열이 동시에 편집될 수 있는 guide RNA construct를 구축하였다. 기존 형질전환법은 소규모 형질전환 시스템에서 캘러스의 재분화 단계에서 결함을 보여주었으나, 선택배지(Selection I, II)와 재분화 배지(Regeneration I)에서 옥신과 사이토키닌의 농도를 수정한 결과 성공적인 옥수수 형질전환을 보여주었다. 특히 Agrobacteria 균주의 효율을 비교하였을 때, LBA4404는 성공적인 형질전환을 보여주었으나, EHA105 균주는 신초분화 과정에서 결함이 나타났다. 이 결과는 소규모 형질전환 시스템에서는 선택배지와 재분화 배지에서의 호르몬 최적화 재조절이 중요한 요건임이 나타낸다. 또한 ZmWUS1 유전자의 프로모터 돌연변이는 하나의 husk leaf 안에 비정상적으로 많은 ears가 발달하는 표현형을 보였고, 이는 ZmWUS1 프로모터 영역이 화서 발달과 진화에 중요한 역할을 맡고 있음을 말해준다. 이번 연구결과는 많은 옥수수 연구자들이 쉽게 옥수수 형질전환체를 활용할 수 있는 매개체가 될 것으로 기대하며, 앞으로 국내 옥수수 연구 발전에 큰 기여가 있을 것으로 기대된다.
Acknowledgments
본 논문은 부산대학교 기본연구지원사업(2년)에 의해 수행되었습니다. 연구비 지원에 감사드립니다.
REFERENCES
-
Ahmadabadi, M., Ruf, S., Bock, R., 2007, A Leaf-based regeneration and transformation system for maize (Zea mays L.), Transgenic Res., 16, 437-448.
[https://doi.org/10.1007/s11248-006-9046-y]

-
Armstrong, C., Green, C., 1985, Establishment and maintenance of friable, embryogenic maize callus and the involvement of L-proline, Planta., 164, 207-214.
[https://doi.org/10.1007/BF00396083]

-
Belhaj, K., Chaparro-Garcia, A., Kamoun, S., Patron, N. J., Nekrasov, V., 2015, Editing plant genomes with CRISPR/Cas9, Curr. Opin. Biotechnol., 32, 76-84.
[https://doi.org/10.1016/j.copbio.2014.11.007]

-
Brooks, C., Nekrasov, V., Lippman, Z. B., Van Eck, J., 2014, Efficient gene editing in tomato in the first generation using the clustered regularly interspaced short palindromic repeats/CRISPR-associated9 system, Plant Physiol., 166, 1292-1297.
[https://doi.org/10.1104/pp.114.247577]

-
Carvalho, C., Bohorova, N., Bordallo, P., Abreu, L., Valicente, F., Bressan, W., Paiva, E., 1997, Type II callus production and plant regeneration in tropical maize genotypes, Plant Cell Rep., 17, 73-76.
[https://doi.org/10.1007/s002990050355]

-
Che, P., Anand, A., Wu, E., Sander, J. D., Simon, M. K., Zhu, W., Sigmund, A. L., Zastrow‐Hayes, G., Miller, M., Liu, D., 2018, Developing a flexible, high‐efficiency Agrobacterium‐mediated sorghum transformation system with broad application, Plant Biotechnol. J., 16, 1388-1395.
[https://doi.org/10.1111/pbi.12879]

-
Chen, Z., Cortes, L., Gallavotti, A., 2023, Genetic dissection of cis-regulatory control of ZmWUSCHEL1 expression by type-B RESPONSE REGULATORS, Plant Physiol., kiad652.
[https://doi.org/10.1093/plphys/kiad652]

-
Cho, M. J., Wu, E., Kwan, J., Yu, M., Banh, J., Linn, W., Anand, A., Li, Z., TeRonde, S., Register, J. C., 2014, Agrobacterium-mediated high-frequency transformation of an elite commercial maize (Zea mays L.) inbred line, Plant Cell Rep., 33, 1767-1777.
[https://doi.org/10.1007/s00299-014-1656-x]

-
Cho, M. A., Park, Y. O., Kim, J. S., Park, K. J., Min, H. K., Liu, J. R., Clemente, T., Choi, P. S., 2005, Production of transgenic maize (Zea mays L.) using Agrobacterium tumefaciens-mediated transformation, J. Plant Biotechnol., 32, 91-95.
[https://doi.org/10.5010/JPB.2005.32.2.091]

-
Doudna, J. A., Charpentier, E., 2014, The new frontier of genome engineering with CRISPR-Cas9, Science, 346, 1258096.
[https://doi.org/10.1126/science.1258096]

-
Fletcher, J. C., 2018, The CLV-WUS stem cell signaling pathway: A Roadmap to crop yield optimization, Plants, 7, 87.
[https://doi.org/10.3390/plants7040087]

-
Frame, B. R., Shou, H., Chikwamba, R. K., Zhang, Z., Xiang, C., Fonger, T. M., Pegg, S. E., Li, B., Nettleton, D. S., Pei, D., Wang, K., 2002, Agrobacterium tumefaciens-mediated transformation of maize embryos using a standard binary vector system, Plant Physiol., 129, 13-22.
[https://doi.org/10.1104/pp.000653]

-
Gelvin, S. B., 2003, Agrobacterium-mediated plant transformation: The biology behind the “gene-jockeying” tool, Microbiol. Mol. Biol. Rev., 67, 16-37.
[https://doi.org/10.1128/MMBR.67.1.16-37.2003]

-
González, G. A., Pacheco, M. G., Oneto, C. D., Etchart, V. J., Kandus, M. V., Salerno, J. C., Eyherabide, G., Presello, D., Lewi, D. M., 2012, Somatic embryogenesis and plant regeneration capacity in Argentinean maize (Zea mays L.) inbred lines, Electron. J. Biotechnol., 15, 9-9.
[https://doi.org/10.2225/vol15-issue1-fulltext-7]

-
Hiei, Y., Ohta, S., Komari, T., Kumashiro, T., 1994, Efficient transformation of rice (Oryza sativa L.) mediated by Agrobacterium and sequence analysis of the boundaries of the T‐DNA, Plant J., 6, 271-282.
[https://doi.org/10.1046/j.1365-313X.1994.6020271.x]

-
Hong, J. K., Lee, G. S., Park, K. J., Kim, J. K., Jang, H. J., Suh, E. J., Kim, K. H., Lee, Y. H., 2019, Production of transgenic maize plants with herbicide resistance through Agrobacterium-mediated transformation, Korean J. Breed. Sci., 51, 290-297.
[https://doi.org/10.9787/KJBS.2019.51.4.290]

-
Hong, J. K., Park, K. J., Lee, G. S., Kim, D. Y., Kim, J. K., Lee, S. B., Suh, E. J., Lee, Y. H., 2017, Callus induction and plant regeneration from immature zygotic embryos of various maize genotypes (Zea mays L.), J. Plant Biotechnol., 44, 49-55.
[https://doi.org/10.5010/JPB.2017.44.1.049]

-
Horstman, A., Bemer, M., Boutilier, K., 2017, A Transcriptional view on somatic embryogenesis, Regen., 4, 201-216.
[https://doi.org/10.1002/reg2.91]

-
Huang, X., Wei, Z., 2005, Successful Agrobacterium-mediated genetic transformation of maize elite inbred lines, Plant Cell Tiss. Organ Cult., 83, 187-200.
[https://doi.org/10.1007/s11240-005-5772-8]

-
Ishida, Y., Hiei, Y., Komari, T., 2007, Agrobacterium-mediated transformation of maize, Nat. Protoc., 2, 1614-1621.
[https://doi.org/10.1038/nprot.2007.241]

-
Ishida, Y., Saito, H., Ohta, S., Hiei, Y., Komari, T., Kumashiro, T., 1996, High efficiency transformation of maize (Zea mays L.) mediated by Agrobacterium tumefaciens, Nat. Biotechnol., 14, 745-750.
[https://doi.org/10.1038/nbt0696-745]

-
Kang, M., Lee, K., Finley, T., Chappell, H., Veena, V., Wang, K., 2022, An Improved Agrobacterium-mediated transformation and genome-editing method for maize inbred B104 using a ternary vector system and immature embryos, Front. Plant Sci., 13, 860971.
[https://doi.org/10.3389/fpls.2022.860971]

-
Kim, H. A., Kwon, S. Y., Yang, M. S., Choi, P. S., 2014, Expression of Dengue virus EIII domain-coding gene in maize as an edible vaccine candidate, J. Plant Biotechnol., 41, 50.
[https://doi.org/10.5010/JPB.2014.41.1.50]

-
Lee, H., Zhang, Z. J., 2016, Maize (Zea mays) Hi‐II transformation via Agrobacterium‐mediated T‐DNA transfer, Curr. Protoc. Plant Biol., 1, 121-137.
[https://doi.org/10.1002/cppb.20016]

-
Lowe, K., La Rota, M., Hoerster, G., Hastings, C., Wang, N., Chamberlin, M., Wu, E., Jones, T., Gordon-Kamm, W., 2018, Rapid genotype “independent” Zea mays L. (maize) transformation via direct somatic embryogenesis, In Vitro Cell. Dev. Biol. Plant, 54, 240-252.
[https://doi.org/10.1007/s11627-018-9905-2]

-
Lowe, K., Wu, E., Wang, N., Hoerster, G., Hastings, C., Cho, M. J., Scelonge, C., Lenderts, B., Chamberlin, M., Cushatt, J., Wang, L., Ryan, L., Khan, T., Chow-Yiu, J., Hua, W., Yu, M., Banh, J., Bao, Z., Brink, K., Igo, E., Rudrappa, B., Shamseer, P. M., Bruce, W., Newman, L., Shen, B., Zheng, P., Bidney, D., Falco, C., Register, J., Zhao, Z. Y., Xu, D., Jones, T., Gordon-Kamm, W., 2016, Morphogenic Regulators Baby boom and Wuschel Improve Monocot Transformation, Plant Cell., 28, 1998-2015.
[https://doi.org/10.1105/tpc.16.00124]

-
Loyola-Vargas, V. M., Ochoa-Alejo, N., 2016, Somatic embryogenesis, An Overview, Springer Int. Publ., 1–8.
[https://doi.org/10.1007/978-3-319-33705-0_1]

-
Mohanty, A., Luo, A., DeBlasio, S., Ling, X., Yang, Y., Tuthill, D. E., Williams, K. E., Hill, D., Zadrozny, T., Chan, A., 2009, Advancing cell biology and functional genomics in maize using fluorescent protein-tagged lines, Plant Physiol., 149, 601-605.
[https://doi.org/10.1104/pp.108.130146]

-
Muppala, S., Gudlavalleti, P. K., Pagidoju, S., Malireddy, K. R., Puligandla, S. K., Dasari, P., 2020, Distinctive response of maize (Zea mays L.) genotypes in vitro with the acceleration of phytohormones, J. Plant Biotechnol., 47, 26-39.
[https://doi.org/10.5010/JPB.2020.47.1.026]

-
Nardmann, J., Werr, W., 2006, The shoot stem cell niche in angiosperms: Expression patterns of WUS orthologues in rice and maize imply major modifications in the course of mono- and dicot evolution, Mol. Biol. Evol., 23, 2492-2504.
[https://doi.org/10.1093/molbev/msl125]

-
Qi, W., Zhu, T., Tian, Z., Li, C., Zhang, W., Song, R., 2016, High-efficiency CRISPR/Cas9 multiplex gene editing using the glycine tRNA-processing system-based strategy in maize, Appl. Biochem., 16, 1-8.
[https://doi.org/10.1186/s12896-016-0289-2]

-
Sahoo, K. K., Tripathi, A. K., Pareek, A., Sopory, S. K., Singla-Pareek, S. L., 2011, An Improved protocol for efficient transformation and regeneration of diverse indica rice cultivars, Plant Methods, 7, 1-11.
[https://doi.org/10.1186/1746-4811-7-49]

-
Sahoo, R. K., Tuteja, N., 2012, Development of Agrobacterium-mediated transformation technology for mature seed-derived callus tissues of indica rice cultivar IR64, GM Crops & Food, 3, 123-128.
[https://doi.org/10.4161/gmcr.20032]

-
Schoof, H., Lenhard, M., Haecker, A., Mayer, K. F., Jürgens, G., Laux, T., 2000, The stem cell population of Arabidopsis shoot meristems is maintained by a regulatory loop between the CLAVATA and WUSCHEL genes, Cell, 100, 635-644.
[https://doi.org/10.1016/S0092-8674(00)80700-X]

-
Shrawat, A. K., Lörz, H., 2006, Agrobacterium‐mediated transformation of cereals: A Promising approach crossing barriers, Plant Biotechnol. J., 4, 575-603.
[https://doi.org/10.1111/j.1467-7652.2006.00209.x]

-
Sidorov, V., Gilbertson, L., Addae, P., Duncan, D., 2006, Agrobacterium-mediated transformation of seedling-derived maize callus, Plant Cell Rep., 25, 320-328.
[https://doi.org/10.1007/s00299-005-0058-5]

-
Songstad, D., Petersen, W., Armstrong, C., 1992, Establishment of friable embryogenic (type II) callus from immature tassels of Zea mays (Poaceae), Am. J. Bot., 79, 761-764.
[https://doi.org/10.1002/j.1537-2197.1992.tb13651.x]

-
Ting, Y., Yu, M., Zheng, W. Z., 1981, Improved anther culture of maize (Zea mays), Plant Sci. Lett., 23, 139-145.
[https://doi.org/10.1016/0304-4211(81)90003-1]

-
Torne, J. M., Santos, M., Pons, A., Blanco, M., 1980, Regeneration of plants from mesocotyl tissue cultures of immature embryos of Zea mays L., Plant Sci. Lett., 17, 339-344.
[https://doi.org/10.1016/0304-4211(80)90166-2]

-
Vega, J. M., Yu, W., Kennon, A. R., Chen, X., Zhang, Z. J., 2008, Improvement of Agrobacterium-mediated transformation in Hi-II maize (Zea mays) using standard binary vectors, Plant Cell Rep., 27, 297-305.
[https://doi.org/10.1007/s00299-007-0463-z]

-
Wang, J., Sun, Y., Li, Y., 2007, Maize (Zea mays) genetic transformation by co‐cultivating germinating seeds with Agrobacterium tumefaciens, Biotechnol., 46, 51-55.
[https://doi.org/10.1042/BA20060065]

-
Werner, T., Motyka, V., Strnad, M., Schmülling, T., 2001, Regulation of plant growth by cytokinin, Proc. Natl. Acad. Sci. U.S.A., 98, 10487- 10492.
[https://doi.org/10.1073/pnas.171304098]

-
Wu, E., Lenderts, B., Glassman, K., Berezowska-Kaniewska, M., Christensen, H., Asmus, T., Zhen, S., Chu, U., Cho, M. J., Zhao, Z. Y., 2014, Optimized Agrobacterium-mediated sorghum transformation protocol and molecular data of transgenic sorghum plants, In Vitro Cell. Dev. Biol. Plant, 50, 9-18.
[https://doi.org/10.1007/s11627-013-9583-z]

-
Xie, K., Minkenberg, B., Yang, Y., 2015, Boosting CRISPR/Cas9 multiplex editing capability with the endogenous tRNA-processing system, Proc. Natl. Acad. Sci. U.S.A., 112, 3570-3575.
[https://doi.org/10.1073/pnas.1420294112]

-
Xie, K., Yang, Y., 2019, A Multiplexed CRISPR/Cas9 editing system based on the endogenous tRNA processing, Plant Genome Editing CRISPR Methods Protoc., 63-73.
[https://doi.org/10.1007/978-1-4939-8991-1_5]

-
Yassitepe, J. E. d. C. T., da Silva, V. C. H., Hernandes-Lopes, J., Dante, R. A., Gerhardt, I. R., Fernandes, F. R., da Silva, P. A., Vieira, L. R., Bonatti, V., Arruda, P., 2021, Maize transformation: From plant material to the release of genetically modified and edited varieties, Front. Plant Sci., 12, 766702.
[https://doi.org/10.3389/fpls.2021.766702]

-
ZHAO, C., ZHANG, L., GE, C., HU, K., 2008, Establishment and optimization of the regeneration system of mature embryos of maize (Zea mays L.), Agric. Sci. China, 7, 1046-1051.
[https://doi.org/10.1016/S1671-2927(08)60145-5]

-
Zhao, Z., Gu, W., Cai, T., Tagliani, L., Hondred, D., Bond, D., Schroeder, S., Rudert, M., Pierce, D., 2002, High throughput genetic transformation mediated by Agrobacterium tumefaciens in maize, Mol. Breed., 8, 323-333.
[https://doi.org/10.1023/A:1015243600325]

Department of Horticultural Bioscience, Pusan National University bije@pusan.ac.kr
Department of Horticultural Bioscience, Pusan National University kangjs@pusan.ac.kr
Department of Horticultural Bioscience, Pusan National Universitykimdaeun@pusan.ac.kr
Department of Horticultural Bioscience, Pusan National Universityk1023@icloud.com